Классификация растровых данных с помощью деревьев решений в R
по адресу http://gis-lab.info/qa/classify-trees-r.html
Пошаговый разбор скрипта для классификации
Статья рассматривает пример классификации с помощью деревьев решений (Classification and Regressions Trees - CART). Программная реализация - R.
Тематическая классификация с помощью деревьев решений состоит из трех основных этапов:
- Подготовка классифицируемых (растровых) данных
- Сбор и подготовка тренировочных данных
- Классификация тренировочных данных - создание модели (дерева)
- Применение модели к классифицируемым данным - получение результата
В этой статье подробно рассматриваются шаги 3 и 4. Чтение и запись растров подробно описывается в отдельной статье, здесь мы останавливаемся только на ключевых моментах. Сбор тренировочных данных полигональными профилями также описывается в отдельной статье, способов собрать такие данные много. В этой статье не рассматривается теория деревьев решений/регрессии.
Из программного обеспечения нам понадобится пакет R для созданяи моделей и классификации. Для R нужно два дополнительных пакета: rgdal для работы с растрами и tree для создания моделей и классификации данных. Также пригодится любая ГИС для визуализации результатов.
Вы можете скачать пакет с исходными, тренировочными и конечными данными использованными в данной статье.
Загружаем пакеты если они еще не загружены:
library(rgdal) library(tree)
Для начала определим некоторые переменные, все они вводятся для удобства:
treesfile = "c:\\temp\\trees.txt"
bnames = c("b11","b12","b13","b14","b15","b21","b22","b23","b24","b25","diff1","diff2","diff3","diff4","diff5")
b2names = c("class",bnames)
numbands = 15
treesfile - текстовый файл, куда будет записываться служебная информация о процессе классификации и результирующая модель - дерево решений. Не обязательно.
bnames - список ("вектор" в терминологии R) строк - названий каналов растра. Обеспечивает легкую идентификацию переменных и лучшую читабельность модели. Не обязательно.
b2names - вектор bnames, в начало которого добавлена строка "class" обозначающая дополнительное поле содержащее значение класса. Не обязательно.
numbands - количество каналов в классифицируемом растре, должно быть также равно количеству полей в файле тренировочных эталонов.
Для служебных целей запишем в treesfile время начала процесса:
sink(file=treesfile) date() sink()
Загрузим и подготовим тренировочные данные, по которым будет создаваться модель:
train="c:\\temp\\train.csv" traindata = matrix(as.integer(scan(file=train)),ncol=numbands+1,byrow=T) class=factor(traindata[,numbands+1]) trainframe=data.frame(class,traindata[,1:numbands]) names(trainframe) = b2names
train - указывает имя и путь к файлу содержащему тренировочные данные. Он представляется из себя простой текстовый файл с разделителями, где каждая запись содержит экстрагированные значения растра во всех каналах (число полей с данными равно numbands). Плюс к данным в файле также есть дополнительное (последнее) поле содержащее значение класса. В нашем случае это 1 (дешифрируемый феномен, сгоревшие территории) или 2 (фон, остальное). Тренировочные данные собираются вручную. Для этого могут использоваться как точечные, так и полигональные профили, как описано например здесь.
Фрагмент файла с тренировочными данными собранными таким образом выглядит так:
17 151 591 358 354 146 138 33 147 161 31971 32013 32558 32211 32193 1 117 151 591 358 354 147 138 42 150 163 31970 32013 32549 32208 32191 1 117 151 591 358 354 142 133 36 146 161 31975 32018 32555 32212 32193 2 113 147 580 354 351 141 131 39 146 161 31972 32016 32541 32208 32190 2
traindata - матрица с числом колонок равным numbands+1 (class), создаваемая в процессе чтения файла с тренировочными данными
class - вектор данных содержащий только значения класса (единицы и двойки)
trainframe - фрейм данных создаваемый небольшой переорганизацией исходных тренировочных данных, класс ставится в начало, тренировочные данные после класса.
в последней операции полям фрейма с тренировочными данными назначаются благозвучные имена заданные ранее. Это позволит при рассмотрении дерева иметь дело с значащими именами а не именами по умолчанию (V1, V2...). Таким образом, если необходимости в изучени самой модели нет, то этот шаг можно опустить.
image = "c:\\temp\\input.tif"
x = new("GDALReadOnlyDataset", image)
width = dim(x)[2]
height = dim(x)[1]
imagedata = data.frame(getRasterTable(x)[3:(numbands+2)])
names(imagedata) = bnames
image - имя и путь к классифицируемому (исходному) растру
x - объект GDAL содержащий указатель на растр
width, height - количество колонок, строк в исходном растре.
imagedata - данные растра, первые два массива getRasterTable(x)[1:2], представляют собой массив координат центров пикселей, широту и долготу соответственно и, таким образом в классификации не участвуют и индекс последнего канала смещается на 2.
Последняя операция опять же делает "красивыми" названия полей во фрейме, так как в исходном растре поля class нет, то применяется bnames, не содержащая class.
numrows=length(trainframe[,1]) indexAll=1:numrows numtrees=30 step=0.5
numrows - количество элементов в тренировочных данных, равно количеству пикселей, т.е. можно было бы также получить умножив ширину на высоту.
indexAll - индекс (последовательность) от 1 до numrows.
numtrees - количество деревьев или сколько раз будет производиться создание модели-дерева по тренировочным данным. Мы собираемся произвести классификацию не один, а numtrees раз, каждый раз выбирая случайную часть набора тренировочных данных и далее усредняя результат, т.н. bagging.
step - какая выборка (доля от 1) тренировочных данных при этом будет выбираться для каждого дерева.
Блок программы выполнящий основную работу выглядит следующим образом:
ans1 = 0
for(i in 1:numtrees) {
sampledata=sample(indexAll,numrows*step,replace=T)
testdata=trainframe[sampledata,]
atree=tree(class ~.,data=testdata,mindev=0,minsize=1)
B=predict.tree(atree,newdata=imagedata,type="vector")
ans1 = ans1 + B[,1]
print(paste("Processing tree#: ",i))
sink(file=treesfile,append = T)
print(summary(atree))
sink()
}
ans1=100*ans1/numtrees
Для каждого из деревьев (которых у нас 30), делается выборка данных sampledata, берется 50% тренировочных данных. На основе этих данных строится дерево решений atree. Потом это дерево применяется к самому снимку. Результат классификации снимка - вектор из 0 и 1, длиной равной количеству пикселей в растре, прибавляется сам к себе. Таким образом, результат, хранящийся в переменной ans1 показывает сколько из 30 деревьев определили пиксель как принадлежащий к классу 1, а сколько к классу 2.
Последняя часть в цикле сбрасывает сами деревья в служебный файл для последующего обучения. Эта информация может быть полезна например для определения, какие переменные (каналы) вносили наибольший вклад в разделение классов. Пример выводимых данных:
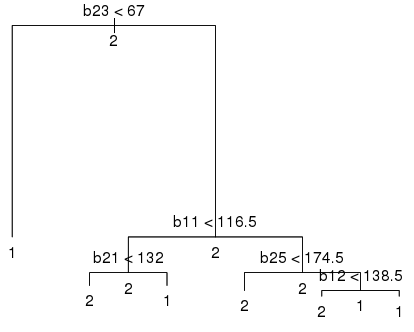
Classification tree: tree(formula = class ~ ., data = testdata, mindev = 0, minsize = 1) Variables actually used in tree construction: [1] "b23" "b11" "diff3" "b12" Number of terminal nodes: 5 Residual mean deviance: 0 = 0 / 393 Misclassification error rate: 0 = 0 / 398
В конце происходит перевод полученных частот в проценты.
Наконец, результат нужно сохранить в выходной растровый файл (output.tif), эта часть подробно описывается в статье "[rgdal.html Работа с растровыми данными в R: rgdal]" и останавливаться подробно на ее разборе мы не будем.
tif_driver <- new("GDALDriver", "GTiff")
tif2 <- new("GDALTransientDataset", tif_driver, height, width, 1, 'Byte')
resmtx = matrix(ans1,width,height)
bnd1 <- putRasterData(tif2, resmtx)
tif_file = "c:\\temp\\output.tif"
saveDataset(tif2, tif_file)
GDAL.close(tif2)
GDAL.close(tif_driver)
Результат получен, теперь нужно загрузить его поверх исходного многоканального изображения и убедиться, что классификация прошла нормально. Разумеется, более точно о качестве классификации можно судить только имея независимый источник для валидации, по которому можно рассчитать ошибки комиссии и омиссии, а также общую точность. Подробнее это рассматривается в статье "Матрица ошибок и расчет показателей точности тематических карт".
Для того, чтобы посмотреть на само дерево классификации воспользуемся следующими командами:
plot(atree) # рисуем структуру дерева text(atree, all=T) # расставляем аннотации
Необходимо также помнить, что, так как наш результат представляет собой диапазон значений от 0 до 100, то перед нами стоит проблема как выбрать порог отсечения. Другими словами, какое число деревьев должно было отнести пиксель к определенному классу, чтобы мы сочли его действительно принадлежащим этому классу. Самая простая стратегия здесь - правило большинства, т.е. пиксель считается относящимся к некоторому классу, если больше чем 50% деревьев отнесло его к этому классу. Конечно, здесь могут быть и более подходящие для ситуации стратегии.

Полную версию скрипта для классификации можно скачать в комплекте с исходными данными.