Консольные инструменты openModeller: различия между версиями
Voltron (обсуждение | вклад) Нет описания правки |
Нет описания правки |
||
| (не показано 5 промежуточных версий 2 участников) | |||
| Строка 1: | Строка 1: | ||
{{Статья| | {{Статья|Опубликована|openmodeller-cmd}} | ||
<p style="text-align:right;">''Чудесной девушке Ане, которая<br />вдохновила меня на завершение статьи.''</p> | <p style="text-align:right;">''Чудесной девушке Ане, которая<br />вдохновила меня на завершение статьи.''</p> | ||
<p class="ann">Описание консольных инструментов openModeller.</p> | <p class="ann">Описание консольных инструментов openModeller.</p> | ||
В [http://gis-lab.info/qa/openmodeller-intro.html предыдущей статье] мы познакомились с openModeller — свободным инструментом моделирования единовременного пространственного распределения с открытым исходным кодом, а также рассмотрели базовые приниципы работы в | В [http://gis-lab.info/qa/openmodeller-intro.html предыдущей статье] мы познакомились с openModeller — свободным инструментом моделирования единовременного пространственного распределения с открытым исходным кодом, а также рассмотрели базовые приниципы работы в графическом интерфейсе openModeller Desktop. Но часто быстрее и проще выполнить необходимые действия воспользовавшись инструментами командной строки. Им и посвящена эта статья. | ||
=== Общие сведения === | === Общие сведения === | ||
| Строка 225: | Строка 225: | ||
#[http://gis-lab.info/qa/openmodeller-intro.html Начало работы с openModeller] | #[http://gis-lab.info/qa/openmodeller-intro.html Начало работы с openModeller] | ||
#[http://gis-lab.info/qa/openmodeller-request.html Описание request-файлов openModeller] | #[http://gis-lab.info/qa/openmodeller-request.html Описание request-файлов openModeller] | ||
#[http://gis-lab.info/qa/openmodeller-demo.html Пример использования openModeller Desktop для новых условий] | |||
#[http://gis-lab.info/qa/gbif-intro.html Глобальная база данных по биоразнообразию — GBIF] | #[http://gis-lab.info/qa/gbif-intro.html Глобальная база данных по биоразнообразию — GBIF] | ||
Текущая версия от 23:16, 28 января 2013
по адресу http://gis-lab.info/qa/openmodeller-cmd.html
Чудесной девушке Ане, которая
вдохновила меня на завершение статьи.
Описание консольных инструментов openModeller.
В предыдущей статье мы познакомились с openModeller — свободным инструментом моделирования единовременного пространственного распределения с открытым исходным кодом, а также рассмотрели базовые приниципы работы в графическом интерфейсе openModeller Desktop. Но часто быстрее и проще выполнить необходимые действия воспользовавшись инструментами командной строки. Им и посвящена эта статья.
Общие сведения
Как уже говорилось в предыдущей статье, openModeller имеет модульную архитектуру: основной функционал предоставляется библиотекой openModeller, алгоритмы загружаются в виде модулей, а для доступа к функциям библиотеки разработано несколько интерфейсов. Одним из первых был реализован консольный интерфейс.
Консольный интерфейс openModeller представлен несколькими приложениями, каждое из которых предназначено для решения своей задачи. Все приложения имеют префикс om_ и на момент написания статьи их было 10:
- om_console
- om_viewer
- om_niche
- om_sampler
- om_model
- om_test
- om_project
- om_algorithm
- om_points
- om_pseudo
Некоторые приложения (а именно, om_viewer и om_niche) зависят от дополнительных компонент и, если в вашей системе необходимые компоненты отсутствуют, могут не запускаться или вообще быть недоступными.
Рассмотрим каждое приложение подробнее. Для демонстрации там, где это возможно, будем использовать тестовый набор данных из поставки openModeller (загрузить архив).
om_console
Консольный фронтэнд к библиотеке openModeller. Используется для создания и применения моделей потенциального распределения. В качестве единственного аргумента принимает request-файл.
om_console request.txt
Развернутое описание request-файла приводится в отдельной статье.
om_viewer
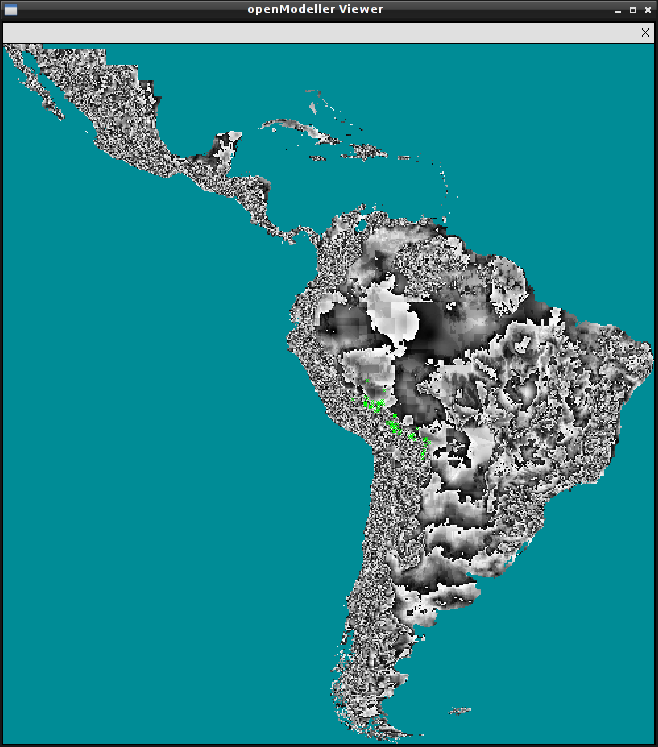
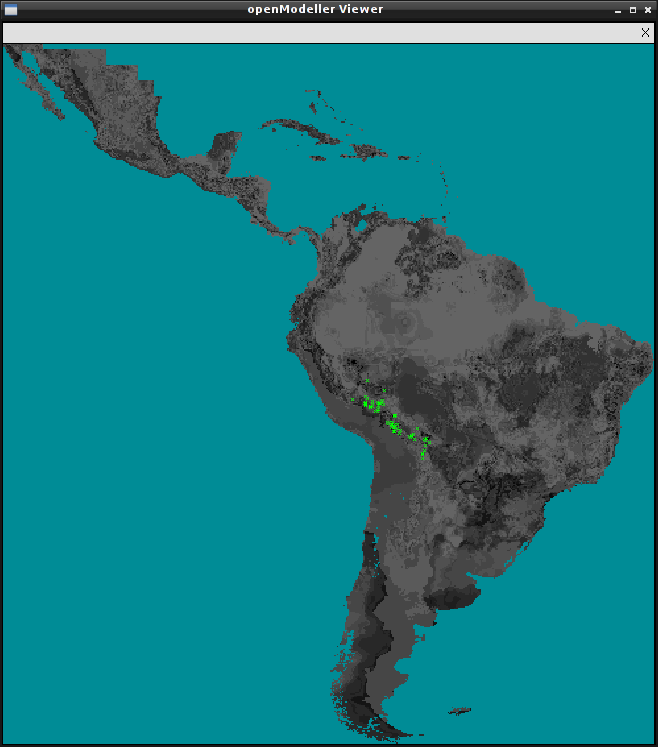
Служит для визуализации слоёв, использующихся при построении модели. В качестве единственного аргумента принимает request-файл.
om_viewer request.txt
Также может использоваться для визуализации итогового распределения. Для активации этого режима служит ключ -r.
om_viewer -r request.txt
Внимание! Приложение доступно только если openModeller собран с поддержкой X11.
om_niche
Выполняет визуализацию сохраненных моделей в экологическом пространстве. Модели в формате XML получаеются в результате работы om_console или om_model.
om_niche -o furcata.xml
На рисунках ниже показаны результаты визуализации ниш, построенных по одним и тем же данным алгоритмами SVM (слева) и MaxEnt (справа).
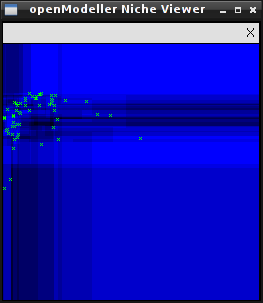
|
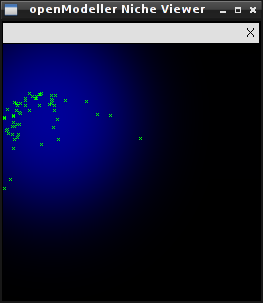
|
Это демонстрационный инструмент, и работает он только в тех случаях, когда эксперимент содержит только два слоя. Главная его цель — показать как разные алгоритмы генерируют разные модели по одним и тем же исходным данным.
Внимание! Приложение доступно только если openModeller собран с поддержкой X11.
om_sampler
В процессе создания модели openModeller извлекает данные (значения пикселей) из каждого слоя, входящего в эксперимент. Набор таких значений для некоторой точки называют «образцом» или «сэмплом» (sample). Именно для получения сэмплов для каждой точки встречи вида и служит это приложение.
om_sampler принимает в качестве аргумента файл, содержащий «ссылки» на климатические и другие слои, а также на файл с информацией о точках встречи. Таким файлом может быть request-файл либо XML-файл с описанием параметров модели, либо XML-файл с сохраненной моделью.
om_sampler --source request.txt
По умолчанию om_sampler печатает на стандартный вывод (т.е. на экран) все данные для каждой точки встречи из исходного файла. Но если указать параметр --dump-env, то выводится будут все данные для каждой ячейки (пикселя) маскирующего слоя. При необходимости можно указать диапазон ячеек используя ключи --cell-start (по умолчанию 0) и --cell-end (по умолчанию 1000). Первой ячейкой считается верхняя левая, обход выполняется слева направо и сверху вниз.
om_sampler --source request.txt --dump-env
В этом режиме вместо координат точек встречи используются координаты центра соответствующей ячейки растра. Если для какой-то ячейки вместо значений выдается «nodata», значит в каком-то из слоёв (включая слой маски) отсутствуют данные.
om_model
Создаёт модель распределения. Исходные данные считываются из XML файла, содержащего параметры в соответствии с описанием элемента ModelParameters. Результирующая модель распределения будет сохранена в другой XML-файл в соответствии с описанием элемента SerializedModel. Необходимо иметь в виду, что каждый алгоритм записывает модель в своем собственном представлении.
При помощи ключа --xml-req задается исходный файл с описанием параметров модели; ключ --model-file задает файл, в который будет записана модель распределения.
om_model --xml-req model_request.xml --model-file acacia_model.xml
Если модель строится для большого числа слоёв или точек встречи можно, при помощи ключа --prog-file, указать файл, в который будет записываться информация о прогрессе операции. Файл прогресса является обычным текстовым файлов, в него в процессе работы приложения могут записываться следующие числа:
- -2 — выполнение прервано
- -1 — находится в очереди
- 0 - 100 — прогресс
Для генерации карты распределения по модели необходимо использовать приложение om_project.
om_test
Инструмент тестирования и проверки моделей. В качестве исходных данных может принимать файл-описание тестов (элемент TestParameters, см. также файл test_request.xml)
om_test --xml-req test_request.xml
или два файла: сохраненную модель и текстовый файл с описанием точек встречи, которые необходимо проверить
om_test --model acacia_model.xml --points test_points.txt --calc-matrix
Примечание: точки должны быть в той же СК и иметь такую же метку, как и тренировочные точки, по которым строилась модель. Кроме того, во время теста будут использоваться те же слои, что использовались при построении модели.
По умолчанию результат работы печатается на стандартный вывод, указав ключ --result можно записать его в файл (формат XML, элемент ModelStatisticsType).
Контролировать процесс тестирования можно при помощи ключей:
- --calc-matrix — рассчитать матрицу ошибок для тренировочных данных
- --threshold — порог вероятности различия точек присутствия и отсутствия при расчете матрицы ошибок
- --calc-roc — рассчитать ROC-кривую для тренировочных данных
- --num-background — число фоновых точек при генерации ROC-кривой в случае если нет точек отсутствия вида
- --max-omission — рассчитать соотношение площадей для точек с максимальным числом пропусков
om_project
Применяет сохраненную модель к некоторому набору слоёв. Результат операции — распределение вероятности в виде растра.
Есть два способа использовать om_project. В первом случае в качестве исходного файла необходимо передать XML файл содержащий настройки процесса применения модели (для примера см. файл projection_request.xml в архиве. Доступно также полное описание элемента ProjectionParameters). В этом случае модель можно применить к набору слоёв, отличному от набора, использовавшегося при построении модели. Примечание: тем не менее, необходимо соответствие между слоями, использовавшимися при создании модели, и слоями, к которым модель применяется.
| Создание модели | Применение модели |
|---|---|
| temperature | temperature_2 |
| rainfall | rainfall_2 |
| precipitation | precipitation_2 |
| и т.д. | и т.д. |
Команда в этом случае выглядит так
om_project --xml-req projection_request.xml --dist-map furcata.img
Ключ --dist-map задает имя выходного файла, в который будет записан результат применения модели (карта распределения вероятности).
Во втором случае om_project использует XML-описание сохраненной модели и применять модель можно только к тем же слоям, которые использовались для её создания. В этом случае также можно указать слой, который будет использоваться в качестве шаблона (--template) и желаемый формат итогового растра (--format).
om_project --model acacia_model.xml --dist-map acacia.img
При необходимости можно указать файл прогресса (--prog-file) и файл статистики (--stat-file).
om_algorithm
Используется для получения информации о доступных алгоритмах. Например, в результате выполнения команды
om_algorithm --list
мы получим список всех доступных алгоритмов в формате «id: название». Получить развернутую информацию о конкретном алгоритме можно вот такой командой
om_algorithm --id algID
где algID — идентификатор нужного алгоритма. А при помощи ключа --dump-xml можно вывести информацию об алгоритмах в XML формате (по умолчанию информация выдается на стандартный вывод, при необходимости используйте перенаправление в файл).
om_points
Служит для загрузки точек встречи из различных источников и их сохранения в текстовом или XML формате.
Загрузка точек встречи возможна из источников для которых существует соответствующий драйвер. В случае необходимости можно создавать свои драйвера для работы с дополнительными источниками данных. Команда
om_points --list
выведет список всех доступных драйверов в формате «id: название (режим доступа)». На момент написания статьи была реализована работа со следующими источниками данных:
- TXT — текст с разделителями. Этот драйвер читает точки из текстовых файлов (разделитель табуляция или запятая) расположенных на локальном диске. В файле должны присутствовать следующие столбцы: идентификатор (id), метка/подпись (label), долгота, широта и (опционально) наличие/отсутствие вида в заданной точке. Этот драйвер всегда доступен и может использоваться для записи данных из других источников. Поддерживается чтение и запись
- XML — описание точек встречи в формате XML. Этот драйвер всегда доступен и может использоваться для записи данных из других источников. Точки встречи хранятся в соответствии с описанием элемента OccurrencesType. Файлы в этом формате также могут использоваться в качестве исходных данных для приложения om_sampler. Поддерживается чтение и запись.
- GBIF — получение данных через GBIF Web Service. Драйвер доступен только если openModeller собран с поддержкой libcurl. В качестве источника данных необходимо указывать http://data.gbif.org/ws/rest/occurrence/list. Доступен только на чтение.
- TerraLib — чтение точек из базы данных TerraLib. Драйвер доступен только если openModeller собран с поддержкой TerraLib. В качестве источника данных необходимо указывать строку подключения в формате
terralib>dbUsername>dbPassword@dbType>dbHost>dbName>dbPort>layerName>tableName>columnName
Доступен только для чтения.
- TAPIR — получение данных через TAPIR Web Service с использованием DarvinCore 1.4 и пространственных расширений. Доступен только если openModeller собран с поддержкой libcurl. В качестве источника данных необходимо указывать точку входа TAPIR. Доступен только для чтения.
Процессом получения точек встречи можно управлять при помощи следующих ключей:
- --source — источник данных. Это может быть путь к файлу на диске (для TXT и XML), URL (для GBIF и TAPIR) или строка подключения (для TerraLib)
- --name — имя (метка) по которому буде выполняться фильтрация точек. Каждая точка в любом источнике имеет имя, что позволяет получать только необходимые данные
- --wkt — система координат точек встречи. По умолчанию широта/долгота, датум WGS84. Полезен только при использовании формата XML, т.к. там есть узел coordinateSystem. Примечание: большинство драйверов предполагает, что точки хранятся с использованием широты/долготы и датума WGS84. Если ключ используется, значение должно быть в формате WKT с экранироваными двойными кавычками
- --type — формат сохранения результатов (TXT или XML). По умолчанию используется TXT
- --split — разбить набор точек на два в заданом соотношении (допустимые значения лежат в диапазоне 0 - 1). Результаты будут сохранены в файлы, заданные парметрами --file1 и --file2. Может быть полезным при создание тренировочного и проверочного наборов
- --file1 — файл в который будет записана первая часть набора в случае разбиения. Обязателен только если используется ключ --split
- --file2 — файл в который будет записана вторая часть набора в случае разбиения. Обязателен только если используется ключ --split
Например, команда
om_points --source http://data.gbif.org/ws/rest/occurrence/list --name "Physalis peruviana"
запросит информацию о точках встречи вида «Physalis peruviana» в базе GBIF и выведет полученные данные на стандартный вывод с использованием формата TXT.
om_pseudo
Используется для генерации случайных точек в пределах заданной географической области (маски). Результат печатается на стандартный вывод в виде текста с разделителями (разделитель табуляция) в совместимом с openModeller виде.
om_pseudo --num-points 20 --mask rain_coolest.tif
Процесс генерации точек контролируется при помощи следующих параметров:
- --num-points — количество точек, которые необходимо сгенерировать
- --mask — файл маски. Это растровый файл в поддерживаемом openModeller формате. Точки не будут генерироваться в ячейках (пикселях), содержащих значение «nodata»
- --label — метка точек, по умолчанию «label»
- --seq-start — целое число, идентификатор первой точки. Последующие точки будут иметь идентификатор на единицу больше, чем у предыдущей. Значение по умолчанию 1
- --proportion — процент точек отсутствия вида. По умолчанию 100 (все сгенерированые точки — точки отсутствия вида)
- --model — файл сохраненной модели. Если этот параметр указан, точки будут генерироваться только в тех ячейках, где модель предсказывает вероятность меньше заданого порога
- --treshold — порог вероятности. Используется вместе с параметром --model. Может принимать значения из диапазона 0 - 1, значение по умолчанию 0.5
- --env-unique — используется вместе с параметром --model, чтобы запретить повторение климатических условий для разных точек. В процессе используются те же слои, по которым строилась модель
- --geo-unique — запрещает создание точек с одинаковыми координатами
Координаты точек всегда создаются с использованием датума WGS84.
Заключение
Как видим, функционал описанных приложений не уступает функционалу графического интерфейса. Консольная природа и более низкое, по сравнению с openModeller Desktop, потребление ресурсов делают их идеальным решением для автоматизированной обработки данных, подбора наиболее оптимального алгоритма, а также для работы с большими объемами информации.